
Contatore di cellule
Al contatore di cellule: Cellule HepG2
Le cellule HepG2 sono una linea cellulare derivata da tessuto epatico di un paziente con carcinoma epatocellulare (Hepatocellular Carcinoma, HCC). Viene spesso utilizzata come sistema modello per l’HCC e per studi sul metabolismo e la tossicità dei farmaci. Le colture sono aderenti, presentano una morfologia epiteliale e tendono a crescere in piccoli aggregati che rendono difficile la conta delle singole cellule. Nelle condizioni di coltura appropriate possono formare superfici apicali e basali distinte, utilizzabili per studi sulle malattie epatiche.
Visualizza il webinar sulla rilevazione di cellule con la tecnologia StainFree
Scarica la nota applicativa sulla rilevazione di cellule con la tecnologia StainFree
Scaricate la nota applicativa sulla rilevazione di cellule con la tecnologia StainFree (Cina)
Scarica l’eBook: Count Cells Like a Pro
Figura 1: Determinazione della percentuale di area coperta con la tecnologia StainFree

Immagini di cellule HepG2 acquisite con il citometro per imaging SpectraMax MiniMax 300. Le cellule sono state identificate utilizzando un’impostazione di analisi personalizzata definita dall’utente. A sinistra sono mostrate le immagini originali in luce trasmessa e a destra sono visibili le stesse immagini con maschere viola che indicano le cellule identificate dal software. Sono mostrate due densità cellulari a confronto.
Figura 2: Analisi StainFree e analisi in fluorescenza della percentuale di area coperta
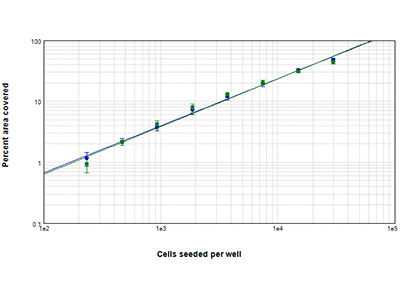
La confluenza delle cellule HepG2 seminate a densità comprese tra 234 e 30.000 cellule per pozzetto è stata analizzata utilizzando la tecnologia StainFree (cerchietti blu) oppure le cellule sono state colorate con il colorante del saggio EarlyTox™ Live Cell, dopodiché è stata analizzata l’area con fluorescenza (cerchietti verdi). I due metodi hanno presentato un elevato tasso di concordanza per quanto riguarda l’area percentuale coperta in una regione di interesse nell’intero intervallo di densità cellulari.
Figura 3: saggio EarlyTox Caspase-3/7 NucView 488
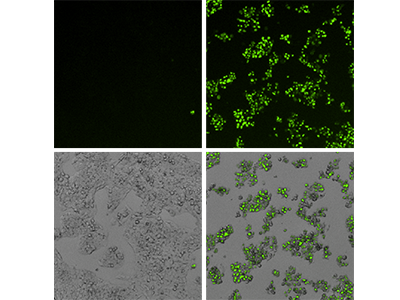
Le cellule HepG2 trattate con un terreno di controllo (pannelli a sinistra) o con capsaisina 200 µM (pannelli a destra) sono state analizzate con il kit del saggio EarlyTox Caspase-3/7 NucView 488 e sottoposte a imaging utilizzando il canale per la fluorescenza verde e quello in luce trasmessa (Transmitted Light, TL) del citometro per imaging SpectraMax MiniMax 300. Fila superiore: immagine in fluorescenza che mostra le cellule apoptotiche marcate con la fluorescenza verde. Fila inferiore: immagini sovrapposte in TL e in fluorescenza che mostrano livelli minimi di apoptosi nelle cellule di controllo e quasi il 100% di apoptosi nelle cellule trattate.
Figura 4: Saggio EarlyTox Caspase-3/7 NucView 488 (andamentto temporale)
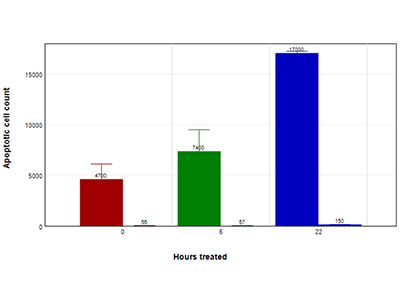
Cellule HepG2 trattate con capsaicina 200 µM (barra sinistra) o con il terreno di controllo (barra destra) per 4,5 ore (barre rosse), 6 ore (barre verdi) o 22 ore (barre blu). L’apoptosi è stata valutata utilizzando il kit del saggio EarlyTox Caspase-3/7 NucView 488. Le cellule sono state incubate con il substrato NucView 488 5 µM e sono state acquisite immagini in corrispondenza dei punti temporali indicati.
Figura 5: Kit del saggio EarlyTox Caspase-3/7 R110 e kit del saggio EarlyTox Glutathione
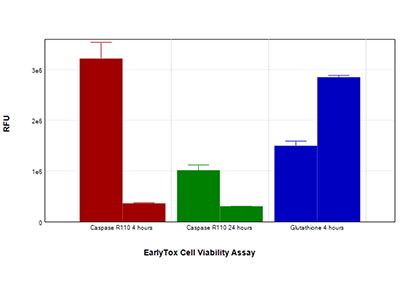
Cellule HepG2 trattate con capsaicina 200 µM (barra sinistra di ciascuna coppia) o con il terreno di controllo (barra destra di ciascuna coppia) sono state analizzate utilizzando il kit del saggio EarlyTox Caspase-3/7 R110 (barre rosse e verdi), che misura l’attività della caspasi, o con il kit del saggio EarlyTox Glutathione (barre blu), che misura la riduzione dei livelli di glutatione, un indicatore precoce dell’apoptosi. Entrambi i saggi sono stati rilevati sul lettore per micropiastre multimodale SpectraMax i3x utilizzando protocolli preconfigurati nel software SoftMax Pro.
Suggerimento: Le cellule HepG2 crescono in aggregati o ammassi di cellule dalla forma irregolare che sono indistinguibili alla semplice osservazione. Un utile metodo per quantificare la crescita o la confluenza delle cellule HepG2 consiste nell’uso della tecnologia StainFree™ per determinare l’area coperta dalle cellule. Le immagini acquisite nel canale TL possono essere facilmente analizzate utilizzando l’impostazione di analisi del campo. È possibile utilizzare l’impostazione di analisi predefinita “Cells” (Cellule) oppure può essere configurata un’analisi personalizzata utilizzando gli strumenti di disegno disponibili nel software SoftMax Pro.
Kit di strumenti per l’analisi delle cellule HepG2
- Piattaforma di rilevazione per micropiastre multimodale SpectraMax ® i3
- Citometro per imaging SpectraMax ® MiniMax™ 300
- Software SoftMax ® Pro
Impostazione dello strumento
Tipo di analisi: analisi del campo
Lunghezza d’onda per l’identificazione di oggetti: TL
Informazione sulla tecnologia di rilevazione cellulare StainFree
Solitamente, i saggi di imaging basati su cellule richiedono l’uso di sonde fluorescenti che possono essere tossiche per le cellule vive o funzionare soltanto in cellule fissate. Un metodo senza marcatura per l’analisi delle conte cellulari e della confluenza cellulare permette ai ricercatori di monitorare in maniera quantitativa la proliferazione e la salute delle cellule senza dover ricorrere a flussi di lavoro laboriosi che potrebbero compromettere la vitalità cellulare.
La piattaforma per micropiastre multimodale SpectraMax i3 con citometro per imaging MiniMax 300 utilizza l’esclusiva tecnologia di rilevazione cellulare StainFree con brevetto depositato per permettervi di eseguire saggi di proliferazione cellulare, di citotossicità e di altro tipo senza coloranti nucleari come il DAPI, che si intercala nel DNA, o coloranti per cellule vive, che a lungo termine sono in realtà tossici per le cellule.