Cos’è l’editing genetico?
L’editing genetico è una manipolazione genetica in cui si procede alla delezione, all’inserimento, alla sostituzione o alla modifica del DNA genomico di un organismo vivente. L’editing genetico è un metodo di targeting sito-specifico per la creazione di tagli nel DNA mediante varie tecniche e non prevede sempre il coinvolgimento di meccanismi di riparazione. È basato su due tecniche: inattivazione e correzione.
L’inattivazione comporta lo “spegnimento” di un gene target, mentre la correzione facilita la riparazione del gene difettoso attraverso un taglio al suo interno. L’editing genetico ha un enorme potenziale in moltissimi campi, tra cui lo sviluppo di farmaci, la chirurgia genetica, i modelli animali, lo studio e il trattamento delle malattie, il settore alimentare, i biocombustibili, la sintesi di biomateriali e altri ancora.
Benché l’uso del sistema CRISPR, una delle principali tecniche di editing genetico, si sia diffuso su larga scala solo di recente, lo studio dell’editing genetico è iniziato alla fine del secolo scorso. Dalla scoperta del sistema CRISPR, la terapia genica, un’ambiziosa strategia precedente, è diventata l’applicazione più ricercata dell’editing genetico. L’obiettivo può essere raggiunto mediante due approcci: l’aggiunta di geni, che comporta un’aggiunta al materiale genetico esistente per compensare geni mancanti o difettosi, e l’editing genetico, che prevede il trattamento di una malattia mediante la modifica diretta del DNA correlato ad essa.
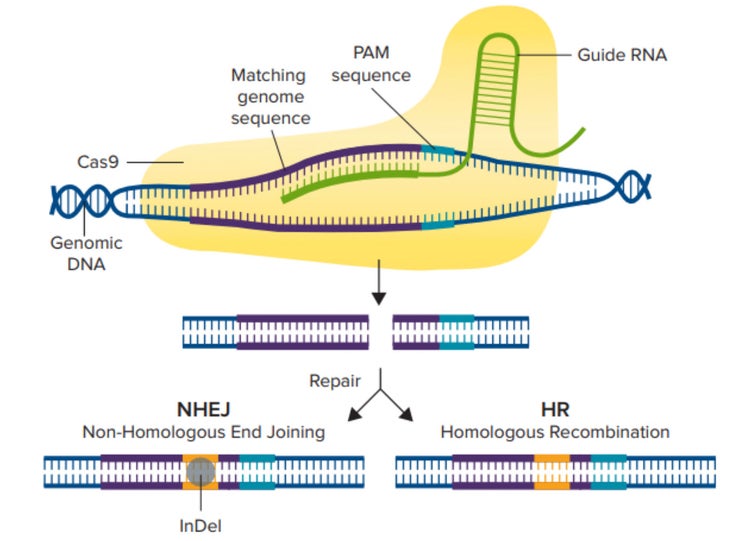
Meccanismo del sistema CRISPR/Cas9. L’enzima Cas9 viene inizialmente attivato mediante il legame a un RNA guida e poi mediante il legame alla sequenza genomica corrispondente che precede immediatamente una sequenza PAM trinucleotidica. Quindi, l’enzima Cas9 crea una rottura a doppio filamento, dopodiché viene utilizzato il meccanismo NHEJ o il meccanismo HDR per riparare il DNA, con conseguente modifica della sequenza genica.
Viene progettato un RNA guida (gRNA), simile a un crRNA, mirato a una regione del gene e l’enzima Cas9 è in grado di causare rotture a doppio filamento in questa regione specifica del genoma della cellula ospite (Figura 1). Dopo che è stata creata una rottura a doppio filamento, la cellula attiva uno dei due meccanismi di riparazione seguenti: il meccanismo di giunzione terminale non omologa (Nonhomologous End Joining, NHEJ) o il meccanismo di ricombinazione omologa (Homology-Directed Recombination, HDR). Il meccanismo NHEJ viene comunemente utilizzato per alterare un gene mediante l’inserimento o la delezione di basi (indel), mentre il meccanismo HDR può essere impiegato per il “knock-in” di un gene reporter o di una sequenza modificata mediante lo scambio di sequenze tra due molecole di DNA simili o identiche.
Ottimizzazione dell’editing genetico con l’ingegnerizzazione basata sulla tecnologia CRISPR
L’acronimo CRISPR significa “Clustered Regularly Interspaced Short Palindromic Repeats”. Queste sequenze di DNA sono state scoperte per la prima volta nel sistema immunitario di procarioti come batteri e archei e hanno acquisito importanza come strumenti di editing genetico a partire dal 2012 (Jinek et al., 2012). Hanno un enorme potenziale in moltissime applicazioni, ad esempio nel campo dell’agricoltura, della modellazione delle malattie, della terapia genica e della scoperta farmacologica, per citarne solo alcune. La precisione di questa tecnologia la rende lo strumento ideale per l’inserimento (knock-in) o la delezione (knockout) e per altre modifiche delle sequenze di DNA. Questa metodica ha sostituito in larga misura i seccanti e costosi strumenti di editing genetico esistenti, come i sistemi TALENS e ZFNS.
Dopo ogni ripetizione palindromica, le sequenze CRISPR contengono tratti di DNA provenienti da precedenti invasori virali e chiamati spaziatori, che contribuiscono alla rilevazione e alla distruzione di virus simili in futuro. La comprensione di questo meccanismo (Jinek et al., 2012) ha portato all’utilizzo del sistema CRISPR prima in cellule eucariotiche (Cong, L, et al., 2013) e successivamente in altri tipi di cellule e organismi in diversi campi. I sistemi CRISPR-Cas9 presentano due componenti principali che formano un complesso ribonucleoproteico. Il primo componente, o RNA guida, si lega a una sequenza di DNA complementare nel genoma e il secondo componente, Cas9 di Streptococcus pyogenes (SpCas9), crea una rottura a doppio filamento in corrispondenza del sito bersaglio. Il legame iniziale della nucleasi avviene in corrispondenza di una sequenza PAM (Protospacer Adjacent Motif), dove si verifica il taglio a monte. Nucleasi CRISPR diverse presentano siti PAM differenti e, una volta che è stato eseguito il taglio, si attiva il sistema di riparazione della cellula e hanno inizio le modifiche del genoma.
Flusso di lavoro dell’editing genetico
Il flusso di lavoro dell’editing genetico basato sui meccanismi CRISPR per ottenere una linea cellulare con modifiche confermate prevede varie fasi. Un’ottimizzazione efficace di queste fasi mediante l’uso di strumenti adeguati contribuisce all’efficienza del processo, permettendo di risparmiare tempo, sforzi e costi necessari per vari progressi scientifici. Questo approccio aiuta ad accelerare le attività di R&S, rivoluzionando la scoperta farmacologica, il trattamento delle malattie, la produzione di colture con modifiche genetiche, ecc. Illustriamo le procedure coinvolte e le soluzioni efficaci che offriamo per sostenere le comunità scientifiche di tutto il mondo nella concretizzazione dei loro sforzi attraverso l’editing genetico.
Soluzioni di ricerca per la validazione delle modifiche genetiche eseguite con la tecnologia CRISPR/Cas9
La famiglia di strumenti Molecular Devices può essere utilizzata in maniera efficace per eseguire/verificare gli esperimenti, garantendo il successo delle procedure di editing genetico. Il nuovo imager CloneSelect Fluorescence (CSI-FL) offre la garanzia di monoclonalità al giorno 0 dopo la dispensazione di singole cellule, permette di verificare l’efficienza di trasfezione e la confluenza cellulare e fornisce dati di screening in fluorescenza multicanale per validare l’efficacia dell’editing genetico con tempi di monitoraggio più brevi, basso rischio di un eccessivo numero di passaggi in coltura e sistemi robotici.
Inoltre, il nostro lettore per micropiastre multimodale SpectraMax i3x può essere utilizzato per valutare l’efficienza di trasfezione, monitorare la crescita cellulare, quantificare DNA e proteine e validare le modifiche effettuate con la tecnologia CRISPR/Cas9 mediante l’analisi con western blot ScanLater. È possibile acquisire immagini di alta qualità degli autofagosomi utilizzando il sistema ImageXpress Micro Confocal, mentre il software HCI MetaXpress è in grado di identificare e quantificare singoli autofagosomi in ogni cellula, permettendo di analizzare le alterazioni fenotipiche derivanti dalle modifiche genetiche effettuate con la tecnologia CRISPR/Cas9.
